染色质在细胞核中折叠成高度复杂的三维结构,并且在细胞分化、增殖等过程中被动态调控。染色质构象捕获技术如5C、Hi-C 和 ChIA-PET 可以捕捉基因组范围的高分辨率染色质相互作用,帮助我们更好地理解三维基因组和生物功能之间的关系。例如,染色质形成的环状结构可以使基因的启动子和相距较远的增强子之间相互作用而影响基因表达。哺乳动物细胞的三维基因组形成约1 Mb的拓扑结构域 (Topologically Associated Domains , TADs),作为基因复制和调控的功能单位。然而由于癌症基因组变异的复杂性,癌症三维基因组的研究目前还较少。
多发性骨髓瘤(Multiple myeloma)是一类浆细胞恶性增殖性疾病,是恶性血液病的一种。多发性骨髓瘤患者在骨髓中多个位点出现浆细胞异常增生,并且伴随着骨骼损伤、免疫力下降等症状,它的发生机制还没有完全被了解。多发性骨髓瘤的基因组主要特征是经常发生染色体拷贝数变异和染色质易位等大尺度的变异。
在这项研究中,李程研究组使用代表两种亚型的骨髓瘤细胞(近二倍体U266和近三倍体RPMI-8226)和正常B细胞(GM12878),对拷贝数变异(CNV)和三维基因组的关系展开研究。通过对这三种细胞的三维基因组测序(Hi-C)、全基因组测序(WGS)和转录组(RNA-seq)数据进行整合分析,发现Hi-C数据的相互作用矩阵受到拷贝数变异的影响,需要进行矫正,同时也表明Hi-C 数据可以用于同时检测三维基因组和基因组拷贝数变异(图1);结合Hi-C和WGS数据可以更精准地发现染色质易位事件(图2);与正常细胞相比,骨髓瘤细胞中TAD的数量增加了25%,TAD的平均长度减小。有意思的是,CNV断点和TAD边界整体上有显著重合(图3),提示两个可能性:这些CNV可能帮助形成新的TAD而影响基因表达、在TAD边界处的CNV断点更容易发生或被癌细胞克隆演化而选择。最后,三维基因组(A/B间隔,TAD)的变化与表达谱变化关联后,能解释部分基因的差异表达,揭示了可能受三维基因组结构影响的骨髓瘤相关基因和通路。
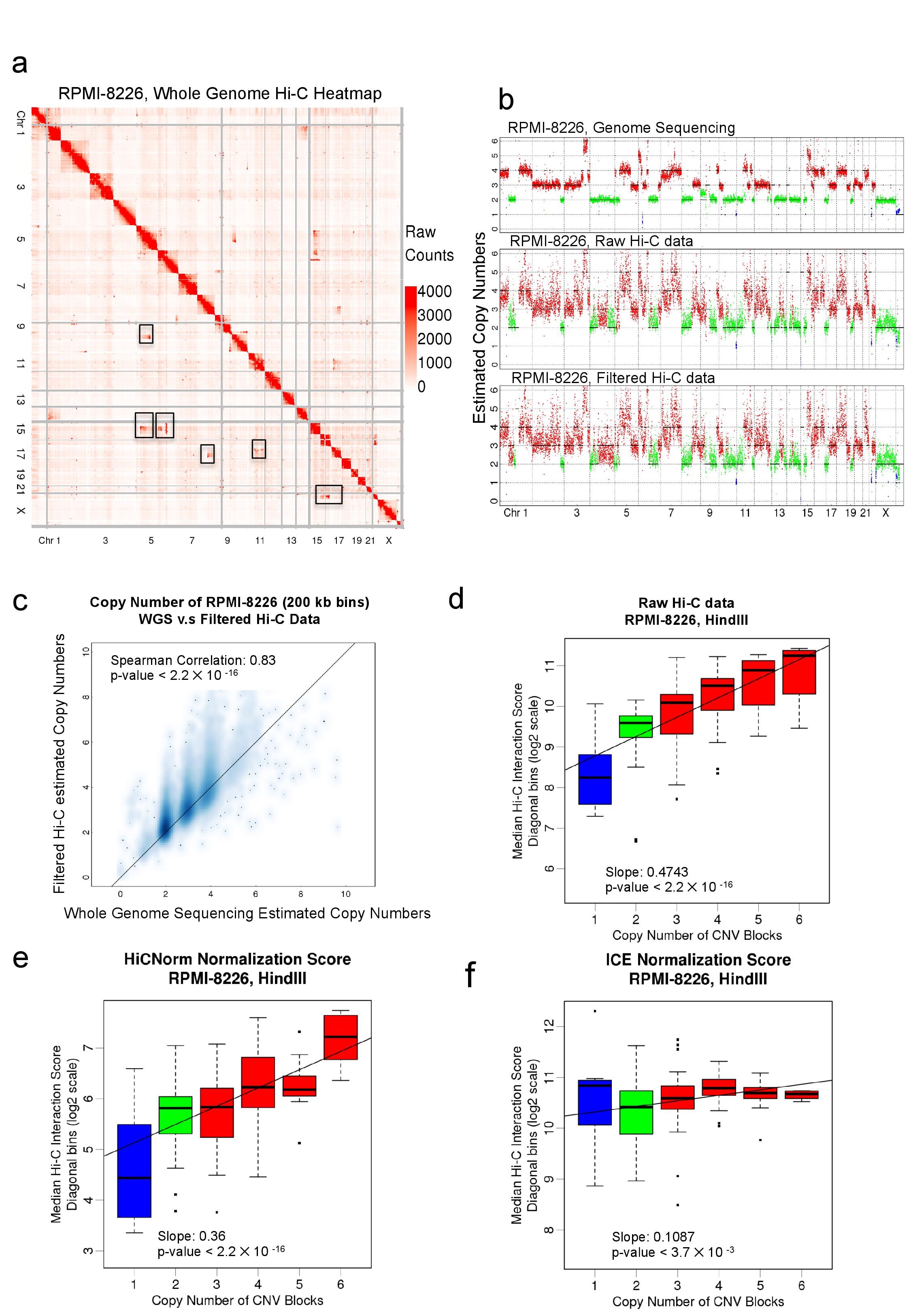
图1:从不同数据类型(WGS, 原始的Hi-C数据和过滤后的Hi-C数据)中得到的基因组拷贝数变异。

图2. Hi-C数据推测的染色质易位事件。
(a) 从WGS数据得到的56个易位事件。(b) Hi-C数据中前100个相互作用最强的染色体间相互作用。
蓝线代表两者之间重合的易位事件。
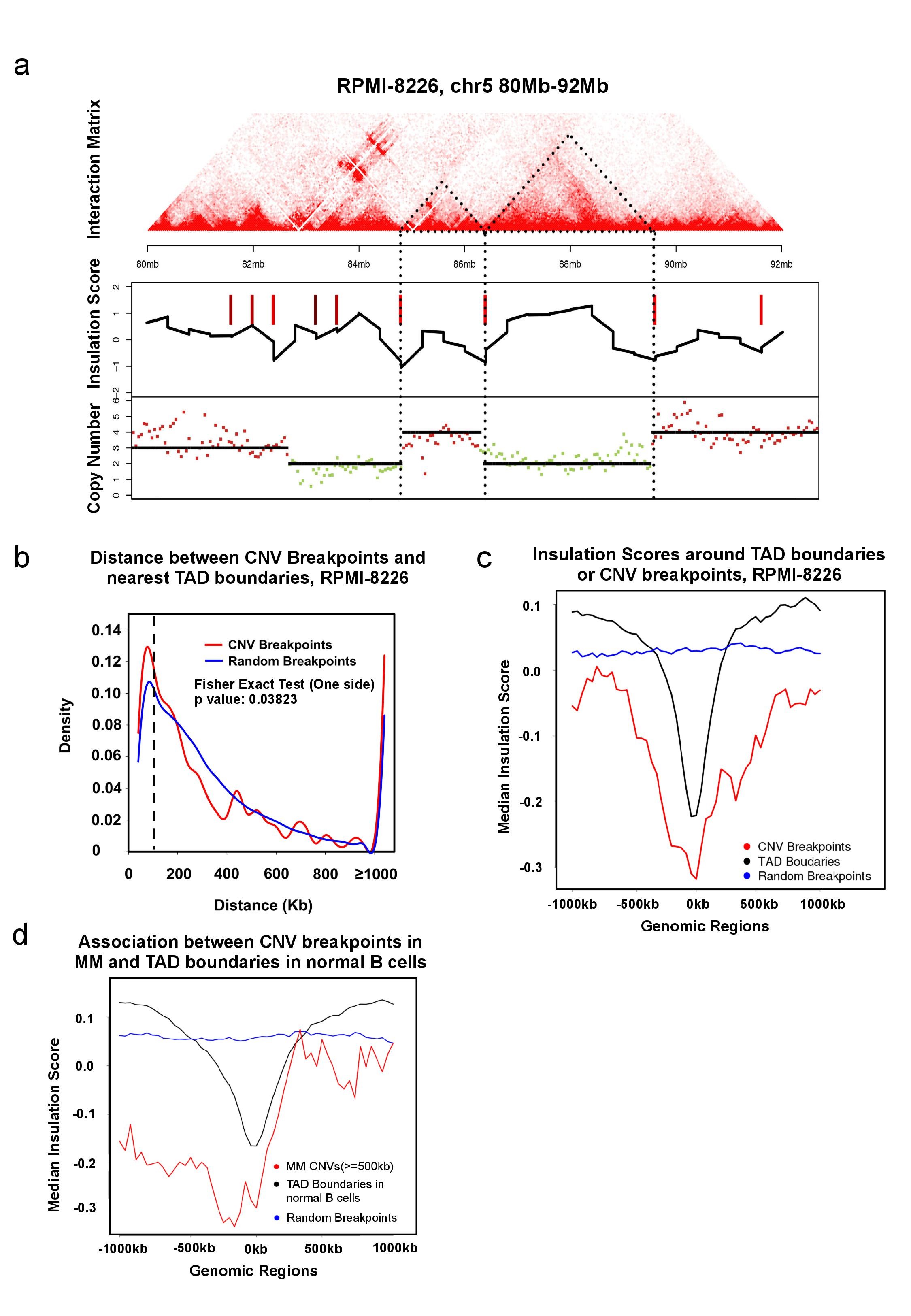
图3. CNV断点和TAD 边界的重合的例子。
第一行是Hi-C得到的染色质相互作用矩阵(经过了拷贝数矫正)。第二行是TAD边界(红色的竖线条)。第三行是从WGS数据得到的拷贝数变化片段。
该研究于2017年12月 5 日以标题“3D genome of multiple myeloma reveals spatial genome disorganization associated with copy number variations”在《Nature Communications》期刊发表。北京大学前沿交叉学科研究院博士生吴朋泽(12级)、李瑞风(14级)和李亭亭博士后为该论文共同第一作者。贾璐萌、朱平、刘一方、陈清、唐岱维、余跃洲参与了部分工作。北京大学高歌、任立晨、徐成冉、孙育杰、清华大学陈阳、高军涛、中国医学科学院黄粤等老师对该工作提供了重要帮助和建议。北京大学生命科学学院、生命科学联合中心李程研究员为该论文的通讯作者。该研究由北大-清华生命科学联合中心、北京大学生命科学学院、科技部、国家自然科学基金资助。

